大規模フィンガープリント類似検索のための簡潔マルチビット木の実装を公開しました
以前のブログで紹介した簡潔マルチビット木のc++による実装 (Succinct Multibit Tree (SMBT)) を公開致しました。ソフトウェアーはgoogle codeからダウンロードすることができます。
http://code.google.com/p/smbt/
SMBTは去年のWABIで発表した内容にもとづいています。
簡潔マルチビット木に関するブログ記事 :
http://d.hatena.ne.jp/tb_yasu/20120808/1344415559
論文 :
Yasuo Tabei: Succinct Multibit Tree: Compact Representation of Multibit Trees by Using Succinct Data Structures in Chemical Fingerprint Searches, Workshop on Algorithms in Bioinformatics (WABI) ALGO, 2012 論文PDFへのリンク
発表スライド :
以下ではSMBTの簡単な使い方を解説します。はじめにソースをダウンロードして解凍してコンパイルします。
tar -xzvf smbt-X.X.X.tar.bz2 cd smbt-X.X.X make
SMBTはフィンガープリントデータベースを入力として、類似度検索のための索引を作成します。このためsmbt-buildコマンドを使用します。
./prog/smbt-built -mode 1 ./dat/fingerprints.dat index
fingerprints.datは入力となるフィンガープリントデータベースのファイル名で、indexは出力の索引ファイルです。
modeは1,2,3から選べます。簡潔マルチビット木を使うためには1または2を、ポインターによるマルチビット木を使うためには3を指定します。この例では1の簡潔マルチビットを使用しています。メモリー使用量は 3 > 2 > 1の順で3のポインターによる実装が最もメモリーを使用し、1の簡潔マルチビット木の実装が最もメモリー効率が良いです。検索速度は 3 > 2 > 1の順で3のポインターによる実装が最も早く、1の簡潔マルチビット木が最も遅くなります。
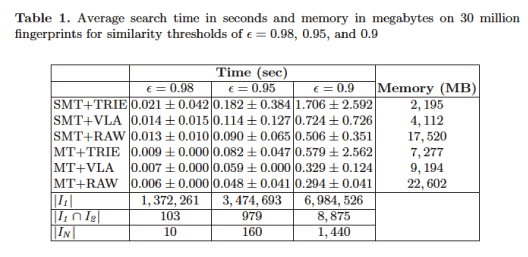
このトレードオフがどの程度かを下のテーブルに示しています(論文からの抜粋)。データはPubChemデータベースから3千万化合物フィンガープリントを用いて、類似度のしきい値0.98, 095, 0.9におけるクエリー1つあたりの検索時間とメモリーのトレードオフを調べています。SMT+TRIEがmode=1, SMT+VLAがmode=2, MT+RAWがmode=3に対応しています。mode=3で, メモリーを20GB消費して検索時間が0.98,0.95,0.9の各しきい値で0.006秒, 0.048秒, 0.294秒、mode=2でメモリーが4GBで検索時間が0.014秒, 0.114秒, 0.724秒, mode=1でメモリーが2GBで検索時間が0.021秒, 0.182秒, 1.7秒程度となります。
注意として、自然言語処理などの単語をフィンガープリントとした場合、単語の種類数が多くなり圧縮が効かなくなる場合があります。その場合はmode=3のポインターによるマルチビット木を使う方が良いです。
作成した索引からクエリーフィンガープリントの類似検索を行うためには、smbt-searchコマンドを使用します。
./prog/smbt-search -similarity 0.8 index ./dat/fingerprints.dat
indexはsmbt-buildコマンドで作成した索引ファイル、fingerprints.datはクエリーフィンガープリントです。 -similarityにて類似度のしきい値を指定します。類似度はJaccard (Tanimoto)類似度です。この例ではしきい値0.8を指定しています。
SMBTの実装では岡野原さんによるrank/select辞書の実装rsdicを使わさせていただいております。
http://code.google.com/p/rsdic/
会議はスロベニアのリュブリャナで開催されました。リュブリャナは町並み気候ともよく楽しめました。会議規模はちょうどよいくらいで、会議の参加者でポストイナ鍾乳洞に行く遠足もありました。